Die Expression eines dominant negativen Mutante innexin in identifizierten Neuronen und Gliazellen reveals_1
EINFÜHRUNG
MATERIALEN UND METHODEN
Versuchstiere
Leech Embryonen in diesen Experimenten verwendet wurde aus einer gemischten Hirudo verbana und Hirudo medicinalis Brutkolonie in unserem Labor erhalten erhalten. Vor der Verwendung wurden sie aus ihren Kokons entfernt und befinden sich in künstlichen Quellwasser (0,5 g L -1 Instant Ocean, Aquarium Systems) bei 22 ° C, und inszeniert nach den Kriterien von Fernandez und Stent (1982). Bei dieser Temperatur Tag 0 (E0) wird als Tag der Kokon Abscheidungs- und Tag 30 (E30) als der Tag des Austritts des juvenile Tier aus dem Kokon definiert. Expression Experimente wurden an Embryonen durchgeführt an den Tagen 12 bis 17 beginnen.
Expressionskonstrukte und Neuronale Expression
Immuncytochemie auf Whole-Mount-Embry
Anti-INX2 Immunzytochemie wurde an ganzen Embryos montiert durchgeführt gegen ein synthetisches Peptidsequenz eines anti-Huhn INX2 polyklonalen Antikörpers unter Verwendung von (YRTKPFVERVEDVESV) in der Nähe des intrazellulären C-Terminus von INX2 und konjugierte an KLH über einen N-terminales Cystein gefunden.
Electrophysiology
Zellkultur
Statistische Analyse
Die Messungen werden in der mittleren gegeben ± Standart Fehler. Die statistischen Signifikanzen für mehr gruppierten Daten wurden durch Einwegvarianzanalyse in Excel, gefolgt von der gepaarten Student-t-Tests bestimmt für Ähnlichkeiten und Unterschiede zwischen den Gruppen zu suchen.
Neuronal Ausgedrückt Tagged Innexine Formular Puncta mit Differential Verteilungen und Größen
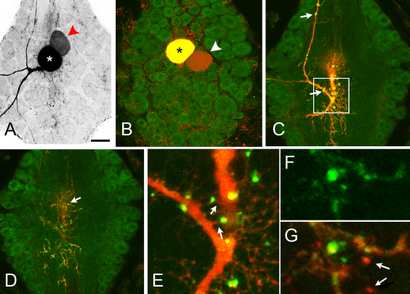
Die Expression von Hve-inx1-EGFP oder Hve-inx2-EGFP ändert nichts an der dorsalen Touch (T) der Zelle farbstoff Ankoppeln an ein Netzwerk von Interneuronen. (A) Neurobiotin Injektion in eine Mitte der Embryonalentwicklung dorsal T sensorisches Neuron (Stern) offenbart, dass es in dem Regel-Farbstoff mit einem Satz von 7-10 klein Inter (red Pfeilspitzen). Die Lage, ist genaue Anzahl und den Grad des Farbstoffdurchganges etwas variabel unter Zubereitungen an diesen embryonalen Stadien. Wenn INX1-EGFP (B, C) oder INX2-EGFP (D, E) wurde in der dorsalen T-Zelle exprimiert, zeigte, Neurobiotin Kennzeichnung eine scheinbar normale Verteilung von kleinen somata Farbstoff-gekoppelter Zellen (weißer Pfeilspitzen in C, E). Beide der markierten Proteine gebildet puncta innerhalb der Dorne der Zellen, obwohl in erster Linie entlang des Hauptprozesses. Man beachte, dass INX2 Plaques erscheinen, größer zu sein, weniger und weiter verbreitet als die von INX1 und viele erschienen angrenzend an Verzweigungspunkten (Pfeile;. Quantifizierung in Fig 3). Bar = 100 um auf.
Eine wichtige Frage, die wir angesprochen wurde, ob der Ausdruck eines markierten Transgen durch eine Zelle, die zum Ausdruck bringt auch die innexin endogen zu anomalen Zelleigenschaften führt. Zuerst verglichen wir die Dorne getaggter-INX1-exprimierenden Neuronen und altersangepassten Kontrollen, wie durch Injektion von Fluoreszenzfarbstoffen oder Koexpression von mCherry offenbart, und hat keine offenkundige Unterschiede in ihrer Größe oder Kennverzweigungsmuster (Daten nicht gezeigt). Zweitens injizierten wir die GJ-Passing-Farbstoff Neurobiotin in die Transgenexprimierenden Neuronen zu fragen, ob dies das Muster der Kopplung an andere Zellen beeinflusst. Für beide Neuronen testeten wir (T, N = 9; Rz, N = 5), der Farbstoff ergab normale Muster der Farbstoff passage [vgl Fig. 1 (A, B) und 2 (A, C, E)].
Wenn Innexin Transgene in Neurons, INX1 und INX14 Puncta Overlap Ausgiebig, Während INX1 und INX6 Puncta Do Not coexprimiert
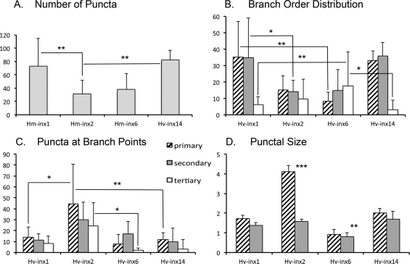
Wir getestet nächste Coexpression von zwei unterschiedlich etikettiert Transgenen in demselben Neuronen die relative Position der beiden Proteine zu untersuchen. Zunächst jedoch ausgedrückt wir INX1-EGFP und INX1-mCherry zusammen zu steuern; mögliche Auswirkungen der Tags, da dies denkbar Hexamer Oligomerisation beeinflussen könnten, und dass das gleiche innexon Protein bestätigen in der Tat innerhalb von Zellen co-lokalisiert. In allen untersuchten Präparate (N = 3) wurden die puncta in den beiden Fluoreszenzkanäle [Fig überlappt beobachtet. 4 (A-D)].
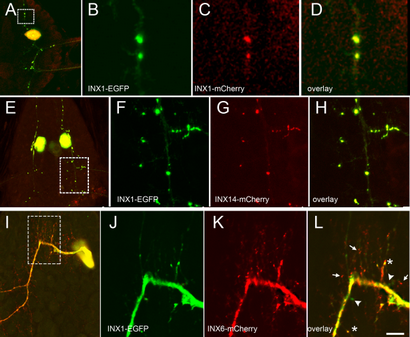
Koexpression von zwei INX Transgene zeigt verschiedene Vorkommen von Kolokalisation in puncta gesamten neuronalen Dorne. (A) geringe Vergrößerung Ansicht eines T-Zell-coexprimieren INX1-EGFP und INX1-mCherry. (B-D) Hohe Vergrößerung Ansichten des eingerahmten Bereich in Tafel A zeigt, die, wenn das gleiche Transgen mit zwei unterschiedlichen fluoreszierenden Markierungen ausgedrückt wurde, Kolokalisation beobachtet wurde. (E) geringe Vergrößerung Ansicht eines Ganglion mit zwei T-Zellen, die jeweils exprimierenden INX1-mCherry und INX14-EGFP. (F-H) Hohe Vergrößerung Ansichten des eingerahmten Bereichs in der Platte E, die zeigen, dass INX1 und 14 co-lokalisiert. (I) geringe Vergrößerung Ansicht einer Anterior Pagoda (AP) Neuron exprimierenden INX1-EGFP und INX6-mCherry. (JL) Hochvergrößerungsansicht des eingerahmten Bereich in Tafel I, nur eine partielle Kolokalisation zeigt, wobei die meisten der INX1 puncta in der Nähe oder innerhalb der Hauptprozesse der Zelle AP konzentriert wird, und der INX6 mehr wird in der gesamten Arbor breit verteilt, insbesondere in die kleineren Terminalprozesse. Sternchen in L zeigen mögliche Beispiele für INX1 und INX6 Kolokalisation, Pfeilspitzen Beispiele für INX1 ohne INX6 und Pfeile Beispiele INX6 ohne INX1 Fluoreszenz anzeigen. Bar = 10 um, mit Ausnahme in den Feldern A, E und I, wo es 100 um auf.
Mutation eines hoch konservierten Prolin zu Leucin Erzeugt ein INX1 Protein mit Dominant Negative Eigenschaften
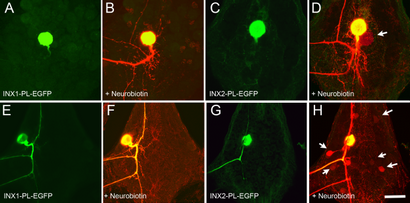
Die Expression von INX1-PL in der Rz und T-Zell-abschafft Farbstoff Kopplung mit einem Homolog davon, aber der Expression von INX2-PL nicht. A. Embryonale Rz Zelle des mutierten Hve-inx1-PL-EGFP Transgen exprimieren. Man beachte, dass das markierte Protein diffus im Zellkörper und Anfangsbereich des Dorns vorhanden ist, aber keine puncta detektiert werden kann. B. gleiche Zelle wie in A, jedoch mit Neurobiotin gefüllt und gefärbt mit Streptavidin-Alexa 568 den Dorn zu visualisieren, die für dieses Embryonalstadium eine normale Anatomie zeigt. Beide Fluoreszenzkanäle angezeigt. Es gibt keine detektierbaren Farbstoffdiffusion zu anderen Neuronen. C. Embryonale Rz Zelle des mutierten Hve-inx2-PL-EGFP Transgen exprimieren. Wie in A wird das markierte Protein hauptsächlich in und in der Nähe des Zellkörpers beobachtet, aber puncta fehlen. D. Wie B, aber in diesem Fall ist die Neurobiotin hat zum Gegen Homolog (Pfeil) diffundiert, was auf anhaltende GJ Kupplung. E. Ein T-Zelle des Hve-inx1-PL-EGFP Transgen exprimieren, versagt auch puncta zu bilden. (F) Neurobiotin Füllung zeigt einen normalen T-Zell Arbor, aber nicht Kopplung mit dem klein Inter zeigen [vgl Fig. 2 (C)]. G. Ausgedrückt INX2-PL-EGFP-Protein auch nicht puncta zu bilden. (H) scheinbar normale Neurobiotin Kopplung mit klein Inter wurde in dieser Zelle (Pfeile) beobachtet. Bar = 75 um auf.
Dass diese Abkopplung Wirkung in Rz und T-Zellen spezifisch ist INX1-PL ist durch seine Abwesenheit angezeigt, wenn das mutierte Glia innexin Hve-inx2 -PL ektopisch in denselben Neuronen exprimiert wurde. Wie das INX1-PL mutiertes Protein, INX2-PL nicht produzierte fluoreszierendes puncta in Neuronen, wenn es in der Rz Zelle exprimiert wurde, aber im Gegensatz zu INX1-PL, normal farbstoff Kopplung mit dem kontralateralen Homolog wurde in Gegenwart von INX2 beobachtet -PL [vgl Fig. 5 (C, D) mit Fig. 1 (A, B)]. In ähnlicher Weise wurde normale T-Zell-Interneuron Farbstoffkopplung beobachtet wird, wenn Hve-inx2 -PL in T-Zellen exprimiert wurde [cf. Fig. 5 (G, H) und 2 (A, C, F)].
Zusammengefasst zeigen diese Ergebnisse, dass INX1-PL in Zellen selektiv als dominant negativen Handlungen, die endogen INX1 (Neuronen) und INX2-PL wirkt selektiv als dominant negativen in Zellen exprimieren, die endogen INX2 (Glia-Zellen) exprimieren.
Inx1-PL Fungiert als Dominant Negative durch die Blockierung der Ausfuhr von INX1 Protein an die Zelloberfläche
Wie oben beschrieben, ausgedrückt Mutante INX1-PL-EGFP Protein diffus im Zellkörper und in proximalen Bereichen des Dorns eines experimentellen Neurons lokalisiert ist, aber es Cluster nicht in puncta. Was passiert mit dem normalen endogenen INX1 dieses Neuron? Da Farbstoffkopplung auch aufgehoben wird, ist eine mögliche Erklärung, dass das mutierte Protein auch die Bildung von puncta durch den normalen innexin blockiert. In Ermangelung eines INX1 Antikörper, waren wir nicht in der Lage, die Verteilung des endogenen INX1 direkt zu beobachten. Stattdessen ausgedrückt wir das normale INX1-mCherry Transgen zusammen mit dem mutierten INX1-PL-EGFP Transgen in verschiedenen Neuronen (N = 3). Wie für das Paar von Neuronen in 7 (A-C) gezeigt ist, werden beiden Gene in markierten Proteine übersetzt, und beide sind mit dem Soma und proximalen Arbor diffus lokalisieren, aber weder Proteinaggregate in puncta überall in der Zelle. Eine parsimonious Erklärung für eine solche Feststellung ist, dass das endogene Protein INX1 auch die Bildung von puncta durch die Mutante verhindert wird, wodurch die Bildung von GJS Verhinderung und zur Aufhebung Farbstoffdiffusion zu den anderen Zellen, mit denen sie normalerweise verbunden sind. Die Beobachtungen oben für INX2-PL-Expression in Gliazellen beschrieben sind mit dieser Erklärung konsistent. Immunostaining mit unseren INX2-spezifischen Antikörpern, die beide, zeigt den Wildtyp und Mutante INX2 Proteine binden, dass die normale INX2 puncta fehlen, wenn INX2-PL [vgl ausgedrückt wird Fig. 6 (B, E)], und Farbstoffkopplung aufgehoben [cf. Fig. 6 (C, F)].
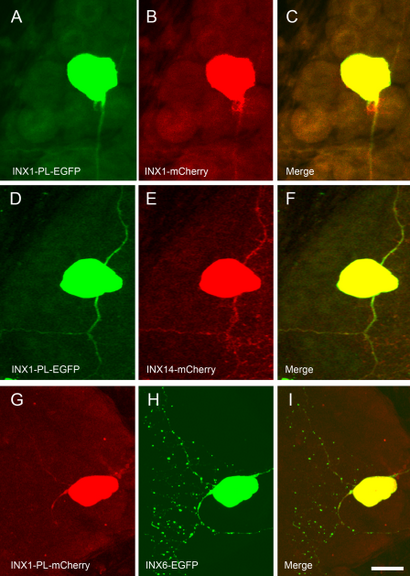
Koexpression von Inx1-PL mit anderen normalen innexin Transgene. (A-C) Coexpression von INX1-PLEGFP (A) und INX1-mCherry (B) in einer T-Zelle (linke Seite) und eine AP-Neuron (rechte Seite) führen zu einer Abwesenheit von INX1 Wildtyp- GJ Proteinen. (D-F) Coexpression von INX1-PL-EGFP (D) und INX14-mCherry in einer T-Zelle beseitigt auch INX14-mCherry puncta in dem Neuron. (GI) Wenn im Gegensatz dazu INX1-PL-mCherry (G) und INX6-EGFP (H) wurden in einer ventralen Druckzelle (PV) ausgedrückt wird, die Wildtyp-INX6 Transgen noch prominent puncta gebildet durch aus dem Dorn des Neurons . Maßstabsbalken = 50 um auf.
Die Expression der INX1-PL Transgene in Zellkultur Zeigt an, dass das INX1-PL Protein nicht an die Zelloberfläche Exportiert wird
Auf der Grundlage dieser Experimente schließen wir, dass das INX1-PL-Mutante GJ Protein exprimiert wird, aber es ist nicht an die Plasmamembran von S2R + Zellen exportiert, sondern erscheinen in internen Gremien zu akkumulieren, vielleicht die Trans-Golgi-Netzwerk der Zellen.
INX1-PL Fungiert als Dominant Negative von INX1 und INX14 aber nicht von INX6
Um zu testen, ob die vorherrschende negative Eigenschaft von INX1-PL nur den Satz von ausgedrückt Innexine auswirkt, die vermutlich mit INX1 interagieren, wir dann für Effekte von INX1-PL auf einem innexin getestet, die nicht ausschließlich mit INX1 nicht colokalisieren, INX6 [siehe Abb. 4 (IL)]. Das Ergebnis der Koexpression INX6-EGFP und INX1-PL-mCherry in einer Reihe von Neuronen war konsequent: die Bildung von puncta von INX6 war nicht betroffen [Abb. 7 (G-I)]. Wir schließen daraus, dass ein PL-Mutante von einer innexin wirkt als dominanter Mutante zu anderen Innexine, die interagieren und colokalisieren mit ihm, aber es funktioniert nicht für Innexine der puncta in anderen Regionen der Laube der gleichen Zelle bilden.
DISKUSSION
Standorte von Innexin Puncta in einem Neuronal Arbor Correlate mit Beteiligung insbesondere Coupled Networks
Expression von spezifischen PL Mutants Entkoppelt Zellen aus ihrem normalen Networks, was darauf hindeutet, dass die normale Zahl von Ausgedrückt Innexine miteinander zusammenwirken
- Die INX1-PL mutierte Protein scheint zum Zellkörper von Blutegel-Zellen und in S2R + Zellen zu konzentrieren scheint nicht an die Zellmembran zu erhalten.
- INX1-PL verhindert auch getaggt INX1-WT-Protein coexprimiert aus dem neuronalen Plasmamembran erreicht und Verbinden oder Plaques bilden. Durch die Erweiterung, nehmen wir an, dass es dasselbe an das endogene Protein INX1 der Fall ist, die durch die experimentellen Neurons mit dem Verlust von Gap Junctions Kupplung passt. Wir können diese vermuteten Störungen direkt beobachten, wenn wir INX2-PL in Gliazellen aufgrund der Verfügbarkeit eines INX2 Antikörper exprimieren: keine endogene INX2 haltigen Plaques nachgewiesen in einer Gliazellen exprimiert, INX2-PL.
- INX1-PL-Expression auch blockiert die Bildung von Plaques durch eine anderes GJ Protein normalerweise in Neuronen exprimiert, INX-14, die in puncta zusammen lokalisiert mit Wildtyp-INX1.
- INX1-PL-Expression beeinflussen nicht coexprimiert getaggt INX6 in Neuronen, die in der Laube des experimentellen Neuron weit verbreitet bildet noch Plaques. INX6 nicht, zum größten Teil, Co-Lokalisierung mit Wildtyp-INX1.
Artikel Infos
Bitte beachten Sie: Wiley-Blackwell ist nicht verantwortlich für den Inhalt oder die Funktionalität von Hintergrundinformationen von den Autoren geliefert. Alle Anfragen (außer fehlenden Inhalten) sollten für den Artikel zu dem entsprechenden Autor gerichtet werden.
LITERATUR
Verwandte Inhalte
Artikel zum einen im Zusammenhang Sie sehen gerade
Unter Berufung auf Literatur
- Anzahl, wie oft zitiert. 7