Die genetische Kartierung und Manipulation Kapitel 8-Dominante Mutationen
Ein gutes Beispiel für ein dominantes Allel in C. elegans ist der rol-6 (su1006) -Allel, das eine Rolle (rol) Phänotyp verursacht. rol-6 (su1006) Tiere, den Rol-Phänotyp zeigen, wenn sie die folgenden Genotypen: rol-6 / rol-6; rol-6 / +; oder + / rol-6. Weil der Rol Phänotyp beobachtet wird, wenn eine einzelne Kopie des mutierten rol-6 (su10060) vorhanden ist, die rol-6 (su1006) Allel dominant sein. Beachten Sie, dass nicht alle Allele eines bestimmten Gens dominant sein wird; gibt es mehrere rol-6-Allele, die rezessive Phänotypen aufweisen. Dominance oder Rezessivität sind Allel-spezifische Eigenschaften. Sie sind nicht die Gen-spezifischen Eigenschaften.
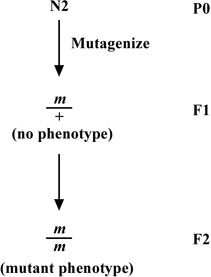
2. Isolierung von dominanten Allele
3. Mapping eine dominante Mutation
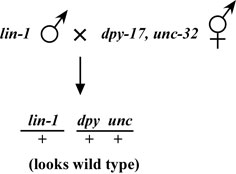
Der Phänotyp einer rezessiven Mutation verschwindet, wenn es in einer Abbildungs Stamm gekreuzt. Betrachten Sie lin-1. was eine multivulval (Muv) Phänotyp, wenn sie in einen Stamm gekreuzt, wie in Abbildung 3 gezeigt, dass wir dann die Co-Segregation der lin-1 punkten und DPY-17 unc-32 in der normalen Weise durch Muv nicht-Dpy UNCs Kommissionier- und unter Hinweis darauf, wie die Dpy Unc Phänotypen oft mit lin-1Muv co-segregieren.
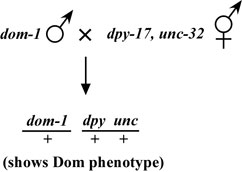
Betrachten wir nun eine dominante Mutation, die, im Gegensatz zu lin-1. weist den mutanten Phänotyps, wenn das Allel als heterozygot ist. Als wir in unsere chromosomalen Mapping-Stämme kreuzen, alle heterozygot Quer Nachkommen den Mutantenphänotyps aufweisen. Lassen Sie sich eine imaginäre dominante Mutation, dom-1 betrachten. wie in 4 gezeigt, die wir Ursachen sagen ein Stachelkopf-Phänotyp. Diese dom-1 / + heterozygot den Stachelkopf Phänotyp zeigen und wird von dom-1 homozygot nicht zu unterscheiden sein. So werden wir nicht in der Lage die Segregation von dom-1 mit dpy-17 punkten. unc-32 durch folgende versetzten Kopf Tiere, weil wir nicht wissen, ob die Tiere dom-1 / + oder dom-1 / dom-1.
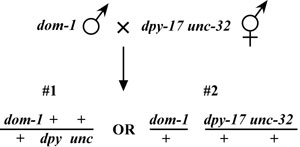
4. Verschiedene Arten von dominanten Mutationen
4.1. Haploinsuffizienz
4.2. Dominant negative Allele
Ein bekanntes Beispiel für ein Gen, das für dominant-negative Mutationen entstehen kann, ist die kleine GTPase Ras. Diese dominant-negative Allele von Ras funktionieren nicht selbst, weil sie bevorzugt GDP binden und im inaktiven Zustand verriegelt bleiben. Darüber hinaus verhindern sie auch den Faktor Ras Austausch (die Ras-GDP bindet und katalysiert GDP / GTP Austausch und die anschließende Aktivierung von Ras) aus Einwirken auf Wildtyp-Ras, im Wesentlichen alle Ras-Aktivität zu töten.
4.3. Dominant gain-of-function (GOF) Allele
4.4. Semi-dominante Allele
Es ist eigentlich ganz typisch für dominante Allele in einem teilweise dominant zu verhalten. Allelen sind halb dominant bezeichnet, wenn die Penetranz des Phänotyps in heterozygoten Tieren (dom-1 / +) geringer ist als die für homozygote Tiere beobachtet (dom-1 / dom-1). Für dom-1. Dies wäre der Fall, wenn dom-1 / dom-1 Tier 100 ist% Stachelkopf und dom-1 / + Tiere 60% Stachelkopf waren. Dies ist ein wichtiger Punkt, da die grundlegenden Kartierungsstrategien oben umrissen wurden 100% Dominanz annimmt. In der Praxis ist dies nicht unbedingt so schwierig zu behandeln, da die Anwesenheit der Mutation wird immer von der nächsten Generation zu sehen ist, wie es der Fall für jede rezessive Allel ist. So Abbilden eine semi-dominante Mutation einfach folgende Nachkommen benötigen für eine zusätzliche Generation zwischen dom-1 / + und + / + Tieren zu unterscheiden.
5. Genetische Tests für Dominanz Klassen
Um zu versuchen, zwischen verschiedenen Klassen von dominanten Mutationen zu unterscheiden, kann eine Anzahl von genetischen Tests durchgeführt werden. Zum Beispiel, um zu bestimmen, ob eine Mutante in einem heterozygoten Tiere beobachteten Phänotyp aufgrund Haploinsuffizienz ist, kann man direkt an Tiere untersuchen, die für einen chromosomalen Defekt heterozygot sind, die das gesamte Gen (wie auch eine Reihe anderer Gene vermutlich) entfernt. Alternativ kann, wenn eine Löschung oder Nullallel des Gens vorhanden ist, ordnet diese Mutation über das Wildtyp-Chromosom könnte eine noch sauberere Antwort. Darüber hinaus Haploinsuffizienz Effekte von hypermorphic Mutationen zu unterscheiden, kann man vergleichen weiter homozygot mutierten Tiere (dom-1 / dom-1) mit den Tieren, die für die Mutation heterozygot sind und der Mangel (dom-1 / df). Wenn die homozygote Mutanten einen schwereren Phänotyp als das mutierte Allel über den Mangel zeigen, dann ist es wahrscheinlich, dass die Mutation zumindest teilweise dominant ist, obwohl man sowohl Dominanz und haploinsufficient Effekte für das gleiche Allel haben kann.
Abschließend verweisen wir die Leserschaft auf eine Reihe von veröffentlichten Arbeiten mit verschiedenen Arten von dominanten Mutationen in C. elegans zu tun (siehe unten). Wir hoffen, dass Sie diese Diskussion von dominanten Allele genossen. Jetzt an die Arbeit zurück, dammit (A. Spencer).