PUMAdb Einzelwertzerlegung Hilfe
Verwandte Hilfe Dokumente
- Repository-Hilfe. Erklärung der PUMAdb des Systems, damit Sie Dateien in verschiedenen Stadien der Analyse speichern und gemeinsam nutzen
- Dateiformate. Informationen über preclustering (.pcl) gruppierte Datentabelle (CDT), Gene Baum (.gtr) und Array-Baum (.atr) Dateien in dem Prozess der Clustering-Daten erzeugt
Sie können SVD verwenden, indem Sie auf der „SVD“ Taste für jede .pcl Datei in Ihrem Repository klicken. Es gibt Hilfedokumente vorgesehen sowohl das Repository und Dateiformate.
Um SVD zu verwenden, wie sie im Zusammenhang mit der Datenbank implementiert ist, werden Sie zunächst eine preclustering (.pcl) Datei in Ihrem Repository setzen müssen. Obwohl das SVD eines Datensatz von der Reihenfolge der Gene und Arrays in den Daten unabhängig ist, könnte eine sinnvolle Reihenfolge Sie dominant eigengenes mit experimentellen Artefakte korrelieren helfen, die auf den Daten oder mit biologischen Prozessen überlagert werden, die in den Daten vorhanden sind. Aus diesem Grund ist es oft von Vorteil, Ihre Arrays mit Hilfe eines Experiments eingestellt oder Array zu bestellen Liste (wie man vielleicht schon für eine Zeitreihe getan) oder durch Gene und / oder Arrays Clustering die Datenbank-Clustering-Pipeline und dann das Abrufen von Daten mit dieser Reihenfolge.
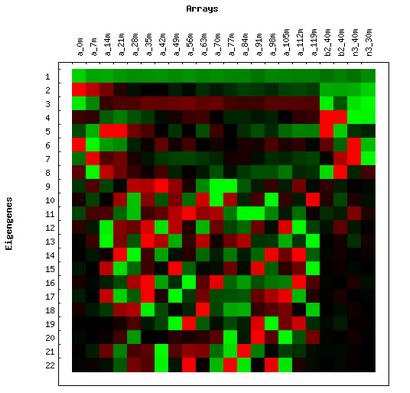
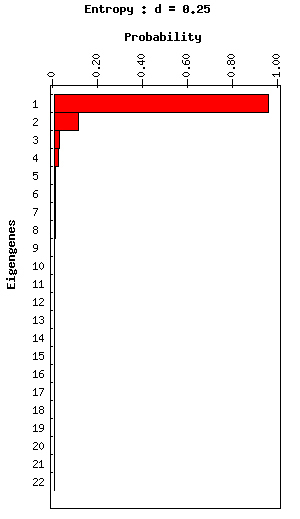
Abbildung 1. Rasteranzeige des Eigengenes (links) und Balkendiagramm Anzeige der
Wahrscheinlichkeiten von Eigenexpression (rechts) von Non-Centered Yeast Cell Cycle Daten
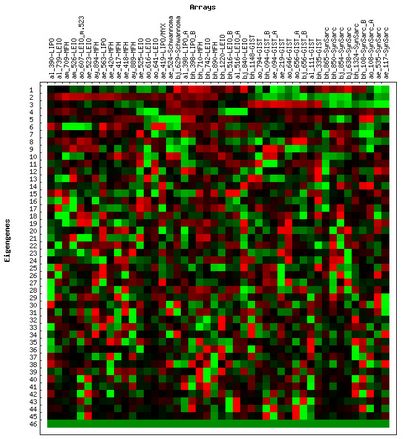
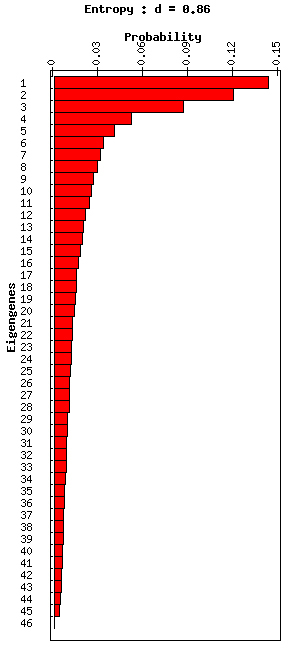
Abbildung 2. Rasteranzeige des Eigengenes (links) und Balkendiagramm Anzeige der
Wahrscheinlichkeiten von Eigenexpression (rechts) von Centered Menschen Sarkom Tumordaten
Sobald eine .pcl Datei erstellt wurde und gespeichert haben, können Sie SVD verwenden auf der „SVD“ -Button in Ihrem Repository, indem Sie auf.
Die Software zeigt die eigengenes Matrix (die am weitesten links stehenden Matrix in 3) in einer roten und grünen Rasteranzeige neben einer Balkendiagrammanzeige ihrer entsprechenden Wahrscheinlichkeiten eigenexpression.
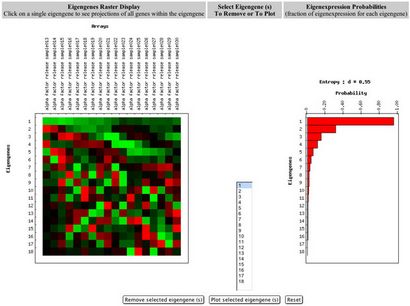
Abbildung 3. Datenbank Werkzeug zum Betrachten und SVD Verwendung in Genexpressionsdatenanalyse.
Jede Zeile in der Rasteranzeige Eigengenes stellt ein eigengene Expressionsmuster. Die oberste Zeile in der Matrix eigengenes ist die erste eigengene, das ist diejenige, die am meisten zu dem gesamten Datensatz trägt. In diesem Display gibt es mehrere Optionen, die unten erläutert:
- Grundstück ausgewählt eigengene (n)
- Anzeigen der Projektion von Genen in jedem eigengene
- Entfernen Sie ausgewählt eigengene (n)
Jede Zeile in dem Balkendiagramm (auf der rechten Seite von Figur 3) stellt die Wahrscheinlichkeit der entsprechenden eigenexpression eigengene (und eigenarray). Zum Beispiel ist die erste (oberste) Stange in der Tabelle die Wahrscheinlichkeit eigenexpression den ersten eigengene (und auch die ersten eigenarray). Es gibt mehr Informationen über die Wahrscheinlichkeit von eigenexpression und Entropie später in diesem Dokument.
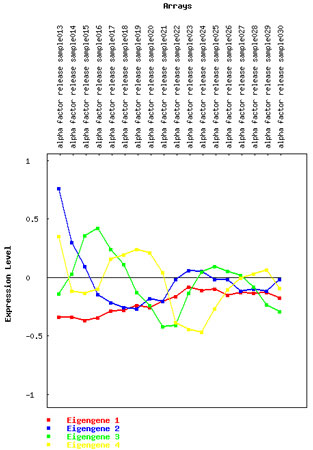
Abbildung 4. Plot das Verhalten von vier eigengenes zeigt.
Klicke auf einen des eigengenes (Zeilen) in der Rasteranzeige der eigengenes Matrix die sortierten Projektionen aller Gene des Datensatz mit diesem eigengene anzuzeigen. Diese Anzeige ermöglicht es Ihnen, die beiden Untergruppen von Genen aus dem Datensatz mit der größten positiven oder negativer Projektion auf die ausgewählte eigengene zu wählen, so dass Sie sie weiter analysieren. Diese Anzeige ist in 5 dargestellt.
Verwenden Sie die „Auswählen von Subsets von Daten“ Daten aus, die Sie in Ihrem Repository als .pcl Datei eingeben oder einfach auf Ihrem Desktop-Computer herunterladen. Vergessen Sie nicht, dass Sie Gene auswählen möchten, die hohe Vorsprünge in sowohl positiver als auch in negativer Richtung. Anschließend können Sie Cluster oder ferner die Daten für diese Gene nach Mustern suchen analysieren. Es gibt mehr Informationen über Gene Projektionen später in diesem Dokument.
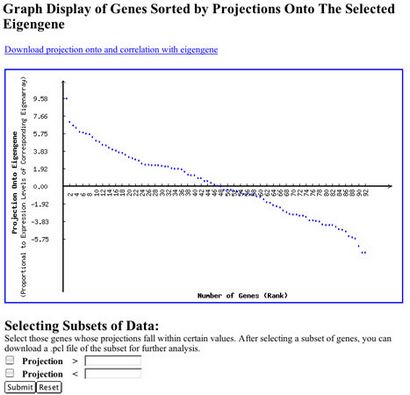
Abbildung 5. Projection von Genen innerhalb einer eigengene. Dieses Bild zeigt, wie alle Gene in einem Datensatz auf eine gegebene eigengene projiziert werden. Dies ist eine Möglichkeit, jene Gene, deren Expression wesentlich dazu beigetragen, durch einen eigengene zu bestimmen.
ein eigengene Entfernen ist mathemetically entspricht den eigenexpression Pegel dieses eigengene (und zugleich, sein entsprechendes eigenarray) zum Einstellen auf Null. Die drei Matrizen (die eigenarrays Matrix, die eigenexpression Matrix und die Matrix eigengene) werden dann wieder mulitplied einen Mikroarray-Datensatz mit den Auswirkungen der eigengene (und seiner entsprechenden eigenarray) entfernt zu rekonstruieren.
eine eigengene Nach dem Entfernen wird die Gene und Arrays, die in dem ursprünglichen Mikroarray-Datensatz waren, sind immer noch da, aber der Ausdruck Daten selbst geändert haben. Zum Beispiel kann ein Gen, das die größten Teil seiner Beteiligung eines gefilterten eigengene hätte erscheint einen nahezu konstanten Null Ausdruck über alle Arrays haben. Da die Datenwerte nun geändert werden, wenn der Filter auf Datenwerte basierend auf Ihre Analyse wichtig sind, möchten Sie vielleicht Ihre rekonstruierten Daten erneut filtern.
Abbildung 6. Schematische (und mathematisch ungenau!) Darstellung, wie SVD findet die „Ansicht“ von Daten, die die größte Varianz erfasst.
Diese Artefakte können dann aus den Gesamtexpressionsdaten entfernt werden (ohne dass die Daten für irgendwelche der Gene oder Arrays zu Verwerfen), indem die entsprechenden eigengene Ausfiltern (n) und eigenarray (s). Diese SVD Datennormalisierung, wo Additiv und gegebenenfalls auch multiplikative experimentelle Artefakte erkannt und ausgefiltert werden, ermöglicht eine bessere weitere Analyse mit Methoden wie hierarchische Clustern, die auf das Vorhandensein eines Artefakts empfindlich sind, die auf den Daten überlagert wird.
Zwei weitere Beispiele dafür sind unten dargestellt:
Die Datenmatrix Ausdruck tabellarisch die Expression beider Gene und Arrays: Die Zeilen der Datenmatrix, die die Expression Expression jedes Gens in dem Datensatz über alle Arrays tabellieren und die Spalten # 8211 die Expression jedes Array auf allen Genen.
Die Eigenexpression Levels
Die W-Matrix hat N x N Abmessungen und die Eigenexpression Level Matrix genannt. Jeder Eintrag zeichnet die Expression eines eigengene in einem eigenarray. Mit anderen Worten, es gibt einen Koeffizienten für die Rolle eines eigengene spielt in einer eigenarray (und umgekehrt). Der erste Eintrag in der Matrix eigenexpression Ebenen erfasst die Höhe der Expression des ersten eigengene im ersten eigenarray.
Jede eigenexpression Ebene stellt die Dominanz des entsprechenden eigengene und eigenarray: Je höher die eigenexpression Ebene, der diese dominante eigengene ist und in den Daten eigenarray. Dies bedeutet zum Beispiel, dass die erste und eigengene eigenarray ist immer dominanter als oder gleich wie dominant als zweite eigengene und eigenarray.
Die Wahrscheinlichkeiten von eigenexpression, die in diesem Balkendiagramm dargestellt sind, werden aus den eigenexpression Ebenen berechnet, das in der diagonalen Matrix des SVD des Datensatz angezeigt werden (siehe Abbildung 8). Die Wahrscheinlichkeit der eigenexpression zeigt die Bedeutung eines eigengene und ihrer in Bezug auf den Anteil der Gesamtexpressionsinformation entspricht eigenarray, dass sie in der Datenmenge erfassen. Es kann als die Wahrscheinlichkeit angenommen werden, dass dieses Muster eigengene als Bestandteil der Expression von einem der Gene manifest ist. Zur gleichen Zeit kann sie auch gedacht werden als die Wahrscheinlichkeit, dass das entsprechende Muster eigenarray manifestiert als Bestandteil der Expression von einem des Arrays. Wahrscheinlichkeiten werden durch Dividieren des Quadrats des l-ten Eigenwert (von Matrix W) durch die Summe der Quadrate aller Eigenwerte berechnet, und sie werden als rote Balkendiagramm auf der rechten Seite in Abbildung 1 dargestellt.
Software stellt die „Entropie“ des Datensatzes in der Beschriftung des Balkendiagramms (siehe zB Figuren 1-3 oben). Die Entropie des Datensatzes misst die Komplexität der Daten aus der Verteilung des Gesamtausdrucks zwischen den verschiedenen eigengenes und entsprechenden eigenarrays und wird aus den Wahrscheinlichkeiten von eigenexpression des Datensatzes berechnet. Die Entropie eines geordneten und redundanter Datensatz, in dem alle die Expression von einem einzigen eigengene und seiner entsprechenden eigenarray erfaßt wird, ist 1. die Entropie eines ungeordneten und Zufallsdatensatz, in dem alle eigengenes und eigenarrays gleichermaßen exprimiert werden, ist 0. üblicherweise die Entropie eines nicht-zentrierten Ausdruck-Datensatz ist etwa 0,1-0,3, und die Entropie eines zentrierten Ausdruck Datensatz ist etwa 0,75 bis 0,95. Filtering eigengene und eigenarray Muster aus dem Datensatz wird die Entropie des Datensatzes ändern.
Die letzte der drei Matrizen, die von SVD ist die Matrix U. Es gibt M Zeilen für Gene, die in dem Originaldatensatz der Daten enthalten (die erste Zeile entspricht das erste Gen im Datensatz entspricht die zweite Zeile zu dem zweiten Gen in der Datenmenge, etc.). Jede Spalte stellt einen eigen-Array (die erste Spalte zu der ersten eigengene entspricht, entspricht die zweite Spalte das zweite eigengene, etc.). Jede Zelle in der Matrix gibt die Koeffizienten, mit denen das Produkt aus W x V T soll, multipliziert wird, um die Menge zu erhalten, die die eigengene für dieses Gen zu dem Datenvektor trägt. Welches ist eigentlich nur der lange Weg neu zu formulieren, dass unser ursprünglicher Datensatz dargestellt werden kann als:
Was bedeutet Überlagerung von Expressionsdaten Mean?
Im Rahmen der SVD, die Expression jedes Gens und Anordnung ist eine Überlagerung (oder eine gewichtete Summe) aller der eigengenes und eigenarrays, respectively. Sie könnten der mathematischen Trennung des SVD denken Sie an die Expressionsmuster der Gene und Arrays in eigengenes und entsprechenden eigenarrays jeweils als Versuch, den Gesamtausdruck Signal in seine erzeugenden Komponenten zu entwirren: unabhängige experimentelle und biologische Prozesse und die entsprechenden Zellzustände. Mit anderen Worten kann SVD verwendet werden, um zu versuchen Expressionsdaten als das Ergebnis eines einfachen Netzwerks zu beschreiben, wo ein paar unabhängigen Quellen des Ausdrucks, experimentellen oder biologische, die Gene, alle im Datensatz beeinflussen (Abbildung 10).
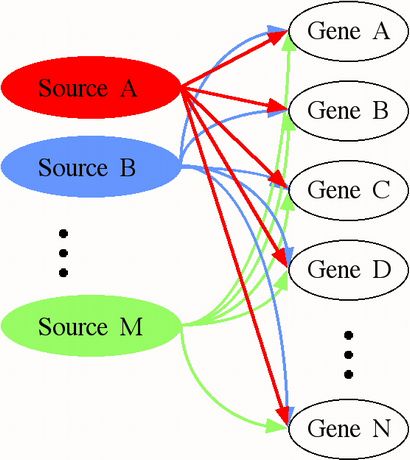
Abbildung 10. SVD kann in einem Versuch verwendet werden, um die Ausdrucksdaten als das Ergebnis eines Netzes von Prozessen zu beschreiben. Jede „Quelle“ stellt einen Prozess (entweder biologische oder artifactual), die eine Wirkung auf die Expression jedes Gens aufweist. Die Wirkung eines jeden Prozesses kann groß oder klein, positiv oder negativ sein.
Gene Projektionen auf und Korrelationen mit ausgewählten Eigengene.
Man beachte, dass die Projektion eines Gens auf eine eigengene relativ zu der eines anderen Gens in der eigenarray aufgeführt ist, die zu dieser eigengene entspricht. Dies bedeutet, dass die Projektion aller Gene in der Datenmenge auf ein eigengene zu dem entsprechenden eigenarray linear proportional ist.
Die Ähnlichkeit eines Expressionsmuster des Gens das Expressionsmuster des eigengene durch „das Gen die Korrelation mit dem eigengene“ (Figur 11) gemessen. Sie können zwischen dem Gen in geometrischen Bedingungen wie der Kosinus des Winkels dieser Korrelation denken und die eigengene, die jeweils einen Vektor im Raum darstellen. Auch hier würden Sie sehen wollen, ob es ein zusammenhängendes biologisches Thema in den Anmerkungen der Gene reflektierte vorhanden ist, mit den größten positiven Korrelationen mit dem eigengene (mit den Mustern am ähnlichsten zu dem dem eigengene). Und Sie würden auch sehen wollen, ob es ein separates kohärentes biologisches Thema in den Anmerkungen der Gene mit den größten negativen Korrelationen (mit den Mustern am ähnlichsten zu den Mustern, die zu dem den eigengene antiparallel ist) reflektierte existiert.
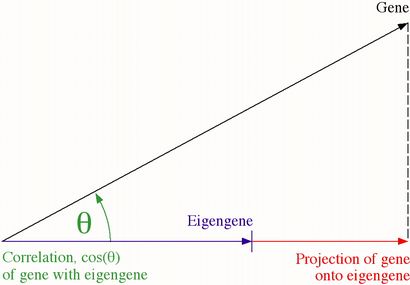
Abbildung 11. Geometrische Beschreibung eines Vorsprunges der Gene Onto und Korrelation mit einem Eigengen
Für diejenigen, die nicht Matrizen seit der High School zu vermehren gehabt haben, ist hier eine sehr kurze Auffrischung, die Sie verstehen, wie Ihre ursprünglichen Daten-Set bezieht sich auf die drei Matrizen, die durch SVD helfen könnten. Wenn wir die beiden Matrizen unter multiplizieren, erhalten wir eine 2 x 2-Matrix wie folgt:
Wir können mit den Zahlen die gleiche Art von Operation tun, wie unten dargestellt:
Wenn wir die drei Matrizen unter multiplizieren, erhalten wir eine 3 durch 2-Matrix wie folgt:
Und mit Zahlen: