Salzbrücke (Protein und supramolekularen)
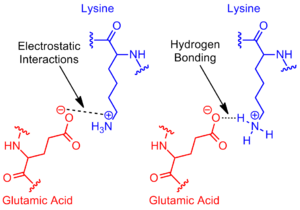
Abbildung 1: Beispiel einer Salzbrücke zwischen den Aminosäuren Glutaminsäure und Lysin demonstriert elektrostatische Wechselwirkung und Wasserstoffbrückenbindung
In der Chemie. eine Salzbrücke ist eine Kombination von zwei nicht-kovalente Wechselwirkungen: Wasserstoffbindungen und elektrostatische Wechselwirkungen (Abbildung 1). Dies wird am häufigsten mit der entropisch ungünstigen gefalteten Konformation von Proteinen beitragen Stabilität beobachtet. Obwohl nicht-kovalente Wechselwirkungen bekannt sind relativ schwache Wechselwirkungen sein, kleine stabilisierende Wechselwirkungen aufsummieren kann einen wichtigen Beitrag zur Gesamtstabilität eines Konformer zu machen. [1] sind nicht nur Salzbrücken in Proteinen gefunden werden, aber sie können auch in der Supramolekularen Chemie zu finden. Die Thermodynamik von jeweils durch experimentelle Verfahren erforscht den freien Energiebeitrag der Salzbrücke zur gesamten freien Energie des Staates zu beurteilen.
Salzbrücken in Proteine bearbeiten gefunden
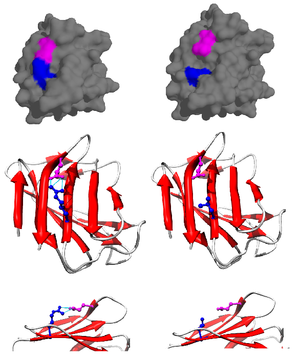
Abbildung 2. Wildtyp- (links) und mutierten (rechts) Form von Lamin A (LMNA, PDB: 1IFR). Normalerweise Arginin 527 (blau) bildet Salzbrücke mit Glutamat 537 (magenta), aber R527L-Mutation führt zu einem Verlust der komplementären negativen Ladung und Struktur Destabilisierung. Auf Phänotyp Ebene dieses Manifest mit überlappenden mandibuloacral Dysplasie und Progerie-Syndrom. [2]
Die Salzbrücke meisten entsteht oft aus dem anionisch carboxylat (RCOO -) entweder Asparaginsäure oder Glutaminsäure und dem kationischen Ammonium (RNH3 +) aus Lysin oder Guanidin (RNHC (NH2) 2 +) von Arginin (Abbildung 2). [1] Obwohl diese die am häufigsten verwendeten, anderen Resten mit ionisierbaren Seitenketten, wie Histidin. Tyrosin. und Serin kann auch teilnehmen, abhängig von äußeren Faktoren Stören ihrer pKa ‚s. Der Abstand zwischen den Resten in der Salzbrücke beteiligt ist ebenfalls als wichtig zitiert. Der Abstand erforderlich ist weniger als 4 Å (400 pm). Aminosäuren größer als dieser Abstand voneinander gilt nicht als eine Salzbrücke zu bilden. [3] Aufgrund der zahlreichen ionisierbaren Seitenketten von Aminosäuren während eines Proteins gefunden Säuren, bei denen der pH-Wert ein Protein platziert ist, um die Stabilität von entscheidender Bedeutung.
Methoden zur Quantifizierung der Salzbrücke Stabilität bearbeiten

Abbildung 3 eine Salzbrücke in T4 Lysozym zwischen Asparaginsäure (Asp) am Rest 70 und ein Histidin (His) an Rest 31
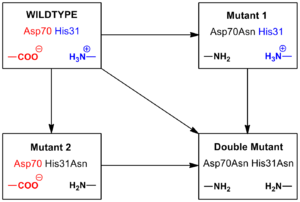
Abbildung 4. Die Mutagenese von T4 Lysozym Salzbrücke zwischen Asp und His 70 31
Nachdem die Mutanten festgelegt sind, können zwei Verfahren eingesetzt werden, um die freie Energie mit einer Salzbrücke verbunden zu berechnen. Ein Verfahren beinhaltet die Beobachtung der Schmelztemperatur des Wildtyp-Proteins gegenüber dem von den drei Mutanten. Die Denaturierung kann durch eine Änderung in Circulardichroismus überwacht werden. Eine Verringerung der Schmelztemperatur zeigt eine Verringerung in der Stabilität. Dies wird durch ein Verfahren quantifiziert beschrieben von Becktel und Schellman wobei die freie Energiedifferenz zwischen den beiden durch & Delta; T & Delta; S berechnet wird. [5] Es gibt einige Probleme mit dieser Berechnung und kann nur mit sehr genauen Daten verwendet werden. [Bearbeiten] In dem T4 Lysozym Beispiel hatte & Delta; S des Pseudo-Wildtypen zuvor bei pH 5,5, so dass die midpoint temperature Differenz von 11 ° C bei diesem pH-Wert durch die berichteten & Delta; S von 360 cal / (mol · K multipliziert berichtet ) (1,5 kJ / (mol · K)) ergibt sich eine Änderung der freien Energie von etwa -4 kcal / mol (-17 kJ / mol). Dieser Wert entspricht die Menge der freien Energie für die Stabilität des Proteins durch die Salzbrücke beigetragen.
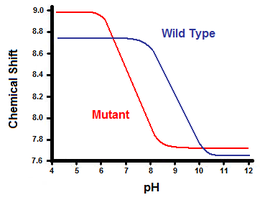
Abbildung 5. Titrationskurve zwischen dem Wildtyp (blau) und der Mutante (rot)
Das zweite Verfahren nutzt magnetische Kernresonanzspektroskopie die freie Energie der Salzbrücke zu berechnen. Eine Titration wird durchgeführt, während die chemische Verschiebung der Aufnahme auf die Protonen des Kohlenstoffs benachbart zu dem Carboxylat oder Ammoniumgruppe entspricht. Der Mittelpunkt der Titrationskurve entspricht den pKa. oder der pH-Wert, wobei das Verhältnis von protonierten: deprotoniert Molekülen beträgt 1: 1. Fortfahrend mit dem T4-Lysozym Beispiel eine Titrationskurve wird durch Beobachtung einer Verschiebung in der C2-Proton von Histidin 31 (Abbildung 5), erhält. Abbildung 5 zeigt die Verschiebung in der Titrationskurve zwischen dem Wildtyp und der Mutante, in der Asp70 Asn. Die Salzbrücke gebildet ist zwischen dem deprotonierten Asp70 und protonierten His31. Diese Interaktion bewirkt, dass die Verschiebung der His31 des pKa gesehen. Im entfalteten Wildtyp-Protein, wo die Salzbrücke nicht vorhanden ist, wird His31 berichtet mit einem pKa von 6,8 in H2 0 Puffer von mäßiger Ionenstärke haben. Figur 5 zeigt einen pKa-Wert des Wildtypen von 9,05. Dieser Unterschied in der pKa wird von den His31 Interaktion mit Asp70 unterstützt. Um die Salzbrücke zu halten, His31 wird versuchen, seine Proton zu halten, so lange wie möglich. Wenn die Salzbrücke gestört ist, wie in der Mutante D70N, verschiebt sich der pKa wieder auf einen Wert von 6,9, viel näher an der His31 im entfalteten Zustand.
Der Unterschied in der pKa kann die Salzbrücke Beitrag zur freien Energie reflektieren quantifiziert werden. Mit Gibbs freie Energie. & Delta; G = -RT ln (Keq), wobei R die universelle Gaskonstante ist, T die Temperatur in Kelvin, und Keq die Gleichgewichtskonstante der Reaktion im Gleichgewicht. Die Deprotonierung von His31 eine Säuregleichgewichtsreaktion mit einem speziellen Keq als die Säuredissoziationskonstante bekannt. Ka. His31-H + ⇌ His31 + H +. Der pKa-Wert wird dann im Zusammenhang mit Ka durch die folgenden: pKa = -log (Ka). Berechnung der freien Energiedifferenz der Mutante und Wildtyp kann jetzt die kostenlose Energie-Gleichung durchgeführt werden, die Definition des pKa. die beobachteten pKa-Werte, und die Beziehung zwischen natürlichen Logarithmen und Logarithmen. In dem T4-Lysozym Beispiel ergab dieser Ansatz einen berechneten Beitrag von etwa 3 kcal / mol zu der gesamten freien Energie. [4] Ein ähnlicher Ansatz kann mit dem anderen Teilnehmer in der Salzbrücke, wie Asp70 im T4 Lysozym Beispiel genommen werden, indem man ihre Verschiebung im pKa nach Mutation des His31 überwachen.